Nat Struct Mol Biol︱王海波课题组揭示I型HDAC复合物Rpd3S识别和催化核小体的机制
组蛋白修饰是调控真核生物基因表达的一种重要的表观遗传机制。最经典的组蛋白修饰之一——赖氨酸的乙酰化(Kac)通常与基因的活跃表达正相关;反之,组蛋白去乙酰化通常导致基因表达的抑制。在活跃表达的基因编码区分布着广泛的组蛋白乙酰化修饰,这些修饰可以中和组蛋白“尾巴”上的正电荷从而减弱其和DNA的相互作用,这种松散的染色质结构有利于RNA聚合酶II(Pol II)在基因上的转录延伸。与此同时,转录行进中的Pol II的磷酸化C端结构域(CTD)直接招募组蛋白甲基转移酶Set2产生H3K36me3修饰[1,2]。H3K36me3修饰作为基因活跃转录区的特征标志(hallmark)会特异性地招募组蛋白去乙酰化酶(HDAC)Rpd3S复合物[3,4],将Pol II转录完的区域里的核小体乙酰化擦除,从而防止其他Pol II在此异常转录(cryptic transcription)。这一精妙的表观遗传调控过程称为Set2-Rpd3S通路,通过不同组蛋白修饰间的相互偶联(crosstalk)实现基因的有序转录。
Rpd3S作为I型 HDAC的原型被广泛研究,它由Rpd3、Sin3、Eaf3、Rco1和Ume1五种亚基组成。Eaf3的Chromo结构域(CHD)提供了Rpd3S与H3K36me3核小体的结合特异性和整体亲和力[5,6],而Rco1的PHD结构域通过优先结合到H3的N端,对去乙酰化酶复合物的功能至关重要[7,8]。有意思的是,Rpd3S更喜欢结合二核小体(di-nucleosome)而不是单核小体(mono-nucleosome)[9],而且结合对核小体间连接DNA的长度有偏好。Rpd3S复合物是如何识别特定二核小体底物相互作用,以及亚基间如何相互协作去除乙酰化修饰仍然是一个谜。
2023年10月5日,浙江大学转化医学研究院/浙江大学医学院附属第二医院/国家基础科学中心王海波课题组在Nature Structural & Molecular Biology期刊上发表了题为“Structure of histone deacetylase complex Rpd3S bound to nucleosome”的研究文章,解析了Rpd3S复合物结合H3K36me3修饰核小体的冷冻电镜结构,并通过生化和酵母遗传学等功能实验,揭示了Rpd3S多价态识别的核小体底物并催化组蛋白去乙酰化的分子机制。

研究组利用化学生物学和生物化学方法在体外重构了带有H3K36me3修饰的二核小体,成功组装了Rpd3S结合二核小体的复合物,通过冷冻电镜技术解析了该复合物的高分辨率结构(图1)。解析的结构清晰地显示Rpd3S整体像一个鳐鱼,从一侧“咬”住单个核小体。有意思的是Rpd3S有2个拷贝的Eaf3亚基,这是之前的研究没有报道过的。
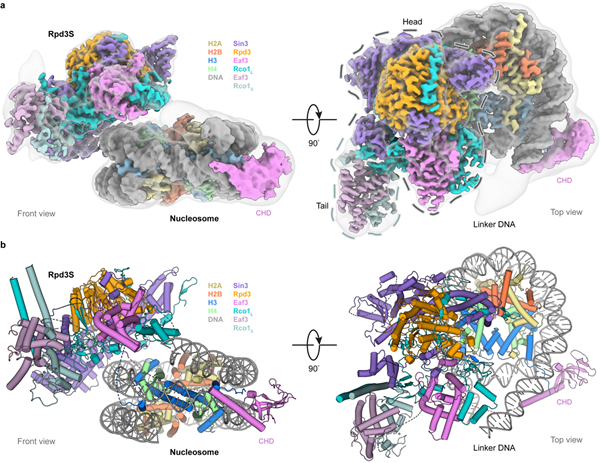
图1. Rpd3S结合带H3K36me3修饰核小体的整体结构
(图源:Li. et al., Nat Struct Mol Biol, 2023)
结构分析显示Sin3亚基作为支架蛋白负责复合物的装配,催化亚基Rpd3位于复合物的中心,Eaf3和Rco1形成稳定的二聚体,是Rpd3S识别核小体底物上特定组蛋白修饰的关键模块。Eaf3的CHD结构域从Rpd3S的主体部分伸展出去,通过芳香笼特异性地识别H3K36me3修饰;Rco1的第一个PHD结构域通过形成β-sheet的方式识别未修饰的组蛋白H3 N末端,并把H3上后续的乙酰化赖氨酸位点递送到催化亚基Rpd3的活性中心(图2)。
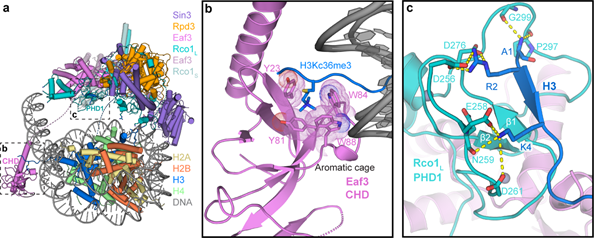
图2. Rpd3S的Eaf3-Rco1组蛋白修饰识别模块
(图源:Li. et al., Nat Struct Mol Biol, 2023)
研究人员随后又用体外酶活实验和酵母遗传学实验研究了Rpd3S对组蛋白H3和H4上乙酰化位点的偏好性以及鉴定了关键相互作用的位点,提出了Rpd3S先去除H3上的乙酰化修饰,再去除H4上的乙酰化修饰的工作模型(图3)。
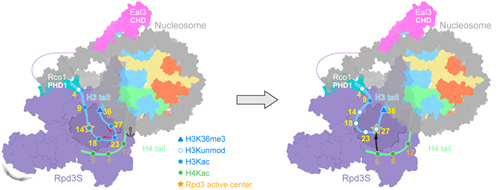
图3. Rpd3S催化核小体去乙酰化的模型
(图源:Li. et al., Nat Struct Mol Biol, 2023)
最后,研究人员利用获得的Rpd3S结合二核小体复合物的中分辨率结构观察到第二个Eaf3-Rco1模块靠近邻近的第二个核小体,提出Rpd3S内在的双Eaf3-Rco1模块使它能同时结合两个核小体,同时也有利于它在染色质上转移去除邻近核小体上的乙酰化修饰(图4)。Eaf3的CHD和MRG结构域之间的长链无规卷曲使得Rpd3S能够容忍一定范围内的连接DNA长度的变化。
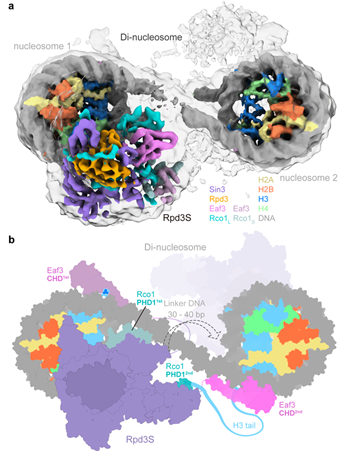
图4. Rpd3S识别二核小体的中分辨率结构和转移模型
(图源:Li. et al., Nat Struct Mol Biol, 2023)
综上所述,该项工作在分子水平上展示了Rpd3S的三维结构及其识别H3K36me3核小体的分子机制,并提出了Rpd3S偏好结合二核小体并在染色质上转移的模型假说。该研究精细地展示了不同组蛋白修饰之间的交互作用和调控机制,为设计特异性的HDAC抑制剂提供了结构基础。值得一提的是,清华大学李海涛/闫创业合作团队于7月19日在Nature杂志在线发表了题为 Diverse modes of H3K36me3-guided nucleosomal deacetylation by Rpd3S 的文章,解析与本研究类似的结构,并通过生化方法对Rpd3S催化H3/H4去乙酰化的构象变化和分子机制做了详尽的探讨。随后,另外3个研究组也在其他杂志发表预印本或者在线文章报道了类似结构,足见研究HDAC及其结合催化底物的机制是目前开发靶向表观遗传因子药物的热点。
王海波研究员是该论文的通讯作者,2023级博士研究生李武龙是论文第一作者,吕志民教授和崔恒军博士为该研究做出了重要贡献。
通讯作者简介
王海波,浙江大学百人计划研究员,博士生导师,浙江大学医学院附属第二医院双聘教授,国家高层次人才青年项目获得者,实验室长期致力于表观遗传和转录调控的结构生物学研究,详情请参考实验室主页(https://person.zju.edu.cn/haibowang#0)。如有兴趣申请王海波实验室的博后、博士研究生、技术员或科研助理,请发Email至:[email protected]。
原文链接:https://www.nature.com/articles/s41594-023-01121-5.
1.Li, J., D. Moazed, and S.P. Gygi, Association of the histone methyltransferase Set2 with RNA polymerase II plays a role in transcription elongation. J Biol Chem, 2002. 277(51): p. 49383-8.
2.Li, B., et al., The Set2 histone methyltransferase functions through the phosphorylated carboxyl-terminal domain of RNA polymerase II. J Biol Chem, 2003. 278(11): p. 8897-903.
3.Carrozza, M.J., et al., Histone H3 methylation by Set2 directs deacetylation of coding regions by Rpd3S to suppress spurious intragenic transcription. Cell, 2005. 123(4): p. 581-92.
4.Keogh, M.C., et al., Cotranscriptional set2 methylation of histone H3 lysine 36 recruits a repressive Rpd3 complex. Cell, 2005. 123(4): p. 593-605.
5.Joshi, A.A. and K. Struhl, Eaf3 chromodomain interaction with methylated H3-K36 links histone deacetylation to Pol II elongation. Mol Cell, 2005. 20(6): p. 971-8.
6.Sun, B., et al., Molecular basis of the interaction of Saccharomyces cerevisiae Eaf3 chromo domain with methylated H3K36. J Biol Chem, 2008. 283(52): p. 36504-12.
7.Li, B., et al., Combined action of PHD and chromo domains directs the Rpd3S HDAC to transcribed chromatin. Science, 2007. 316(5827): p. 1050-4.
8.Ruan, C., et al., Homodimeric PHD Domain-containing Rco1 Subunit Constitutes a Critical Interaction Hub within the Rpd3S Histone Deacetylase Complex. J Biol Chem, 2016. 291(10): p. 5428-38.
9.Huh, J.W., et al., Multivalent di-nucleosome recognition enables the Rpd3S histone deacetylase complex to tolerate decreased H3K36 methylation levels. EMBO J, 2012. 31(17): p. 3564-74.


